LGI1-ADAM22/23経路の破綻は海馬原性のてんかん性脳症を引き起こす
~国際多機関共同研究により病態解明~
2025年08月12日
研究報告
【本研究のポイント】
・国際共同研究を通じて、はじめてのLGI1およびADAM23の両アレル性バリアントを有する発達性てんかん性脳症患者を見出した。
・LGI1両アレル性バリアントを有する患者は、残存するLGI1機能に応じて軽微な行動異常から致死性てんかん発作まで広汎な症状を示すことを見出した。
・Lgi1欠損マウスの解析から、てんかんの焦点は海馬原性であることを突き止めた。
・LGI1-ADAM22/23経路が破綻したマウスの行動解析から、この経路の破綻は、てんかん以外に高次脳機能障害も引きおこすことを見出した。
概要
名古屋大学大学院医学系研究科 神経情報薬理学の深田正紀 教授、分子細胞薬理学の深田優子 准教授、東京大学大学院医学系研究科の平野瑶子 大学院生らの研究グループは、東京大学薬学系研究科の池谷裕二 教授、自然科学研究機構生理学研究所の稲橋宏樹 技術職員、および7ヶ国の遺伝学者、臨床医との共同研究により、“LGI1–ADAM22/23関連疾患スペクトラム”という新しい脳疾患カテゴリーを提案し、その分子、神経回路および個体における病態メカニズムを解明しました。
近年のヒト全ゲノムシーケンス解析やヒトエクソーム解析の普及により、新規の遺伝子バリアント(変異)が次々と報告されていますが、それらの多くの病態メカニズムは未だ十分に明らかにされていません。これまでに本研究グループは、常染色体顕性てんかんの原因遺伝子として報告されていた分泌タンパク質LGI1とその受容体ADAM22/23の研究を進め、LGI1-ADAM22/23複合体が神経細胞のシナプス伝達や可塑性、興奮性制御に重要な役割を果たしていることを報告してきました。本研究では、国際多機関共同研究を通じて、最初のLGI1とADAM23の両アレル性バリアント(変異)を有する「発達性てんかん性脳症患者」を見出しました。これらLGI1バリアントの分泌レベルを高精度に測定したところ、残存するLGI1機能と患者の臨床症状の重篤度の間に高い相関性があることが明らかになりました。また、Lgi1欠損マウスの電気生理学的解析から、てんかんの焦点は海馬原性であることを突き止めました。さらに、LGI1-ADAM22/23経路が破綻したマウスの行動学的解析により、LGI1-ADAM22/23経路の破綻は、てんかん発作以外に認知機能障害も引きおこすことを見出しました。
これらの発見は、 “LGI1–ADAM22/23関連疾患スペクトラム”という新しい脳疾患分類を提案し、その病態メカニズムを明らかにした点で重要であると共に、多くの国際機関との共同研究の成果としても意義深いと言えます。
本研究成果は、2025年8月6日付国際科学雑誌『Brain』誌に掲載されました。
背景
てんかんは、総人口の0.5~1%が発症する最も有病率が高い脳疾患の一つです。てんかんは突発的に生じるけいれん発作や意識消失発作を反復する疾患の総称であり、脳の神経細胞や神経回路の異常興奮により引き起こされます。てんかん原因遺伝子の多くが神経細胞の興奮を担うイオンチャネルをコードしていることから、遺伝学的要因が関与するてんかんを「チャネル病」と捉えることも少なくありません。一方、近年の次世代シークエンサーを用いたエクソーム解析や全ゲノム解析(*1)の進歩により、稀な遺伝性疾患の遺伝子バリアント(*2)が次々と見出されるようになってきました。
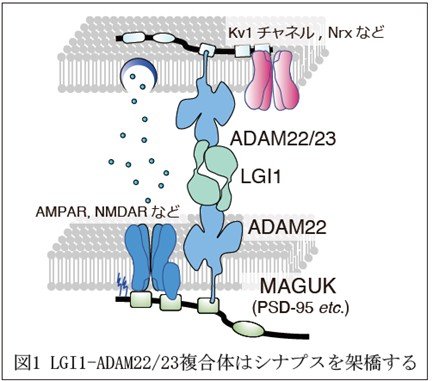
本研究グループは、常染色体顕性外側側頭葉てんかんの原因遺伝子産物LGI1とその受容体ADAM22およびADAM23の機能解析を進めてきました。これまでに、LGI1-ADAM22/23複合体が、(1)シナプス間を架橋し、神経細胞のシナプス伝達や可塑性を制御すること、および(2)軸索起始部に集積し、神経細胞の興奮性制御に重要な役割を果たすことを報告してきました(図1)。そして、2016年〜2023年にかけて、国際共同研究を通じてADAM22の両アレル性バリアント(*2)を有する20名のてんかん性脳症患者を報告しました[Neurol Genet 2016; Brain 2022; Brain Commun 2023]。一方で、LGI1のバリアントは、家族性てんかんでのみ報告されており、てんかん以外の脳病態との関連は不明でした。

研究成果
今回、研究グループは、国際多機関共同研究により、4家系6人の患児において、世界で初めて両アレル性LGI1バリアントを同定しました(図2、3)。患児は全員が、新生児期または乳児期にてんかんを発症し、全般性発達遅滞、および知的発達症を呈しました。また、6人中5人は若くして死亡しました。この重篤な臨床所見は、焦点てんかんのみを呈する、片アレル性LGI1バリアントを有する患者とは対照的でした。さらに、新生児期に致死性てんかん発作を示した、両アレル性ADAM23バリアントを有する最初の症例も見出しました。
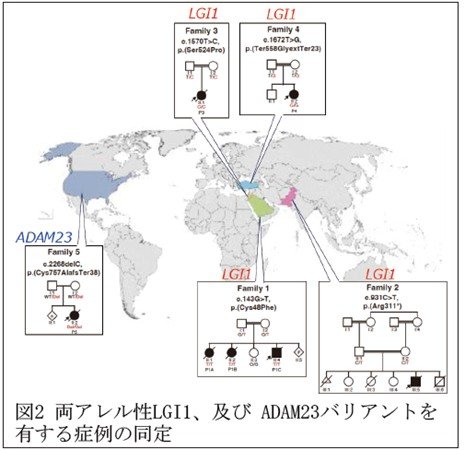
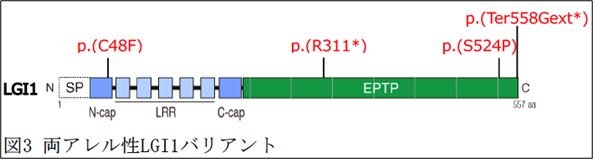


研究グループは、HiBiTシステム(*3)という手法を活用し、LGI1バリアントの分泌量を高精度に定量したところ、今回見出したLGI1バリアントの分泌量は、野生型LGI1と比較して、0.5%、約7%、40%と、異なる程度に減少していることを見出しました。重要なことに、LGI1機能の欠損が大きければ大きいほど、臨床的重症度が高くなることが判明しました。例えばLGI1分泌量がゼロの症例では、患児は生直後に死亡しましたが、分泌量がある程度維持されている症例では軽症の発達遅滞が認められるものの長期間の生存が認められました。つまり、LGI1分泌量の改善、促進は本疾患の治療戦略になりうることが示唆されました。また、LGI1、ADAM22、およびADAM23の両アレル性バリアントを有する患者は、非常に類似した臨床症状を示すことが明らかになりました。
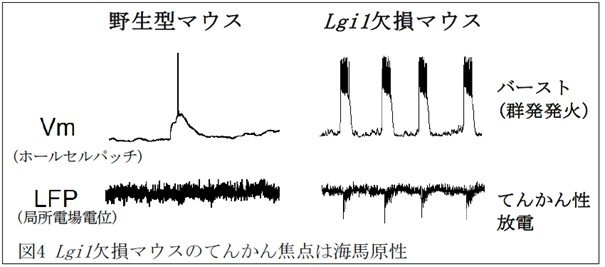
次に、LGI1てんかんの焦点を特定するために、Lgi1 KOマウスを用いた電気生理学的解析を行いました。このマウスでみられる表現型は、上記の分泌量がゼロのLGI1バリアントがおこす病態を模倣すると考えられます。ここでは、脳から海馬組織をまるごと摘出し(in toto 解析)、CA3錐体神経細胞からホールセルパッチ記録を、同時にCA3錐体細胞層から局所電場電位(LFP)を記録しました。Lgi1 KOマウスのCA3ニューロンは、海馬外部からの入力刺激がない状況で、頻繁なバースト(群発)発火を示し、これらのバースト発火はCA3領域のてんかん性放電と同期していました(図4)。以上の結果より、LGI1の機能が欠失した場合のてんかんの焦点は海馬原性であることが示されました。
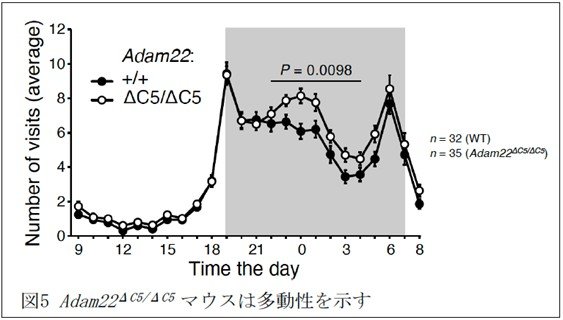
さらに、研究グループは、インテリケージシステムを活用して、LGI1-ADAM22/23経路が破綻した疾患モデルマウス(Adam22ΔC5/ΔC5マウス)の行動学的解析を行いました。インテリケージシステムは最大16匹まで集団飼育が可能な自動行動解析システムで、自然環境下での行動・記憶学習などを測定できます。一連の行動実験の結果、疾患モデルマウスは、野生型マウスに比べて多動であり(図5)、学習・認知機能障害を示すことが明らかになりました。つまり、LGI1-ADAM22/23経路の破綻はてんかんのみならず、脳高次機能にも影響を与えることが明らかになりました。
以上の結果は、ヒトにおいて“LGI1–ADAM22/23関連疾患スペクトラム”という新しい疾患分類を提案し、その病態メカニズムを明らかにした点で重要であると共に、多くの国際機関との共同研究の成果としても意義深いと言えます。
今後の展開
LGI1–ADAM22/23分子経路の破綻は、機能欠損のタイプや程度に応じて、これまで報告されてきた”焦点てんかん”以外に、知的発達症や自閉症状、認知機能障害、致死にいたるまで広汎な脳病態と関連することが分かってきました。したがって、LGI1-ADAM22/23経路の強化・賦活化は、遺伝性てんかんのみならず、様々な神経疾患の治療標的にもなりうることが強く期待されます。
支援・謝辞
ご協力賜りました患者さんとご家族の皆様に心より御礼申し上げます。本研究は、国立研究開発法人日本医療研究開発機構(AMED)の「脳神経科学統合プログラム(個別重点研究課題)」(研究開発課題名:てんかん、知的発達症と自己免疫性神経疾患にまたがる共通病態の解明)、「難治性疾患実用化研究事業 疾患基礎基盤研究プロジェクト」(研究開発課題名:VGKC複合体を標的とする神経疾患の病態解明) 、日 本 学 術 振 興 会 科 学 研 究 費 補 助 金 (22K15208、22K06451、23K23986、23H00374, 23H04243、23K18228)、および武田科学振興財団の支援を受けて行われました。また、本研究は自然科学研究機構 生理学研究所共同利用研究(24NIPS317)の支援を受けて行われました。
用語説明
*1)エクソーム解析、および全ゲノム解析:エクソーム解析は、ゲノムのうちエクソン領域のみを網羅的に解析する手法。一方、全ゲノム解析は、ゲノム全体の塩基配列を決定し、その遺伝情報を総合的に解析する手法。いずれも遺伝性疾患の原因遺伝子の探索や、がんの遺伝子変異解析を目的に活用される。
*2)バリアント:DNA配列の多様性や違いを指す言葉。バリアントには臨床的に病的、臨床的意義不明、良性などに分類される。片アレル性バリアントは、2つあるアレル(対立遺伝子)のうち、片方のアレルのみに存在するバリアントを指す。一方、両アレル性バリアントは2つのアレルの両方にバリアントが存在することをいう。
*3)HiBiTシステム:検出対象タンパク質(今回はLGI1)には11アミノ酸からなるHiBiTタグを付加されており、検出試薬にはナノルシフェラーゼ断片(LgBiT)と基質が含まれている。HiBiTとLgBiTが高い親和性で結合することにより、活性のあるナノルシフェラーゼが再構成される。発光測定により、高感度、高精度でHiBiTタンパク質を定量することができる。
*2)バリアント:DNA配列の多様性や違いを指す言葉。バリアントには臨床的に病的、臨床的意義不明、良性などに分類される。片アレル性バリアントは、2つあるアレル(対立遺伝子)のうち、片方のアレルのみに存在するバリアントを指す。一方、両アレル性バリアントは2つのアレルの両方にバリアントが存在することをいう。
*3)HiBiTシステム:検出対象タンパク質(今回はLGI1)には11アミノ酸からなるHiBiTタグを付加されており、検出試薬にはナノルシフェラーゼ断片(LgBiT)と基質が含まれている。HiBiTとLgBiTが高い親和性で結合することにより、活性のあるナノルシフェラーゼが再構成される。発光測定により、高感度、高精度でHiBiTタンパク質を定量することができる。
論文情報
雑誌名:Brain
論文タイトル:Biallelic LGI1 and ADAM23 variants cause hippocampal epileptic encephalopathy via the LGI1–ADAM22/23 pathway
論文タイトル:Biallelic LGI1 and ADAM23 variants cause hippocampal epileptic encephalopathy via the LGI1–ADAM22/23 pathway
著者:Yoko Hirano,1,2 Yuri Miyazaki,3 Daisuke Ishikawa,4 Hiroki Inahashi,5 Zuhair Nasser Al-Hassnan,6 Giovanni Zifarelli,7 Peter Bauer,7 Javeria Raza Alvi,8 Tipu Sultan,8 Michelle L. Thompson,9 Abdullah Sezer,10 Bahadır Konuşkan,11 Razan S. Hajir,12 Ayman W. El-Hattab,13,14,15 Stephanie Efthymiou,16 Ayuki Ishida,3 Norihiko Yokoi,3 Hans-Christian Kornau,17,18 Dietmar Schmitz,17,18 Harald Prüss,17,19,20 Henry Houlden,16 Yuji Ikegaya,4,21,22 Yuko Fukata,23,† Masaki Fukata3,24,† Reza Maroofian16,†
†These authors contributed equally to this work.
1 Department of Pediatrics, Graduate School of Medicine, The University of Tokyo, Tokyo 113-8655, Japan
2 Department of Pediatrics, The University of Tokyo Hospital, Tokyo 113-8655, Japan
3 Division of Neuropharmacology, Nagoya University Graduate School of Medicine, Nagoya, Aichi 466-8550, Japan
4 Graduate School of Pharmaceutical Sciences, The University of Tokyo, Tokyo 113-0033, Japan
5 Section of Mammalian Transgenesis, Center for Genetic Analysis of Behavior, National Institute for Physiological Sciences, National Institutes of Natural Sciences, Okazaki, Aichi 444-8787, Japan
6 College of Medicine, Alfaisal University, Department of Medical Genomics, Genomic Medicine Centre of Excellence, King Faisal Specialist Hospital & Research Centre, Riyadh 11211, Saudi Arabia
7 CENTOGENE GmbH, 18055 Rostock, Germany
8 Department of Paediatric Neurology, The Children's Hospital and the University of Child Health Sciences, Lahore, Punjab 54600, Pakistan
9 HudsonAlpha Institute for Biotechnology, 601 Genome Way, Huntsville, AL 35806, USA
10 Department of Medical Genetics, Ankara Etlik City Hospital, Ankara 06170, Türkiye
11 Division of Pediatric Neurology, Department of Pediatrics, Ankara Etlik City Hospital, Ankara 06170, Türkiye
12 Research Institute for Medical and Health Sciences, University of Sharjah, Sharjah, United Arab Emirates
13 Department of Clinical Sciences, College of Medicine, University of Sharjah, Sharjah, United Arab Emirates
14 Clinical Genetics, University Hospital Sharjah, Sharjah, United Arab Emirates
15 Genetics and Rare Disease Center, Burjeel Medical City, Abu Dhabi, United Arab Emirates
16 Department of Neuromuscular disorders, UCL Queen Square Institute of Neurology, University College London, London, WC1N 3BG, UK
17 German Center for Neurodegenerative Diseases (DZNE) Berlin, 10117 Berlin, Germany
18 Neuroscience Research Center (NWFZ), Cluster NeuroCure, Charité-Universitätsmedizin Berlin, corporate member of Freie Universität Berlin and Humboldt-Universität zu Berlin, 10117 Berlin, Germany
19 Helmholtz Innovation Lab BaoBab (Brain antibody-omics and B-cell Lab), 10117 Berlin, Germany
20 Department of Neurology and Experimental Neurology, Charité-Universitätsmedizin Berlin, corporate member of Freie Universität Berlin and Humboldt-Universität zu Berlin, 10117 Berlin, Germany
21 Center for Information and Neural Networks, National Institute of Information and Communications Technology, Suita City, Osaka 565-0871, Japan
22 Institute for AI and Beyond, The University of Tokyo, Tokyo 113-0033, Japan
23 Division of Molecular and Cellular Pharmacology, Nagoya University Graduate School of Medicine, Nagoya, Aichi 466-8550, Japan
24 Center for One Medicine Innovative Translational Research (COMIT), Nagoya University, Nagoya, Aichi 464-8601, Japan

