CRISPR/Cas9システムによるゲノム編集は、Talen等の従来方法に比べて簡便であることから、急速に利用が広がっている。最近、ゼブラフィッシュにおいて、CRISPR/Cas9システムを介した長鎖DNA断片のノックインが、相同組み換え非依存的なDNA修復を介して可能であるということが報告された(Auer, et al: Genome Res 24, 142–153)。論文の中で著者らは、トランスジェニックフィッシュ中のGFP配列を、Gal4に変換することに成功している。これまで、ゼブラフィッシュには効率的なノックインの方法がなかったが、この報告によりノックインフィッシュが作製できる可能性が示唆された。しかし、内在遺伝子領域に長鎖DNA断片をノックインした報告は未だなく、CRISPR/Cas9システムを用いて実用的な効率でノックイントランスジェニックフィッシュを作製できるかについては不明であった。そこで、本研究において我々は、Auer等の方法を改変したうえで、細胞種特異的にレポーター遺伝子を発現する複数系統のノックインゼブラフィッシュの作製を試みた。
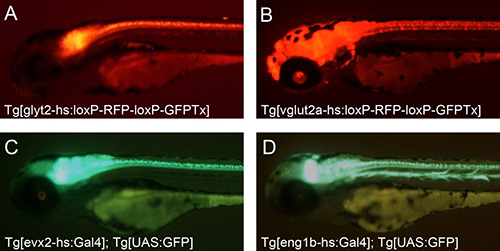
ノックインフィッシュを作製する際には、ゲノムの目的配列を標的とするsgRNA1と、ドナープラスミドを標的とするsgRNA2、熱ショックプロモーターを含むドナープラスミド、Cas9 mRNAを受精卵に注入した(図1)。効率の良いsgRNAを用いた場合には、5-10%の注入胚で、レポーター遺伝子の発現が、内在性遺伝子の発現領域において幅広く観察された。これらの胚を選択して育てたところ、25%以上という高い効率で次世代にノックインフィッシュを得ることができた。本研究では、4種類の内在性ゲノム配列を標的としたノックインを行い、それぞれ、特定の神経細胞群にレポーター遺伝子を発現するノックインフィッシュ系統を複数得ることに成功した(図2)。
本研究は、CRISPR/Cas9システムを用いて、レポーター遺伝子を内在性ゲノム配列にノックインしたトランスジェニックフィッシュ作製の初めての報告である。CRISPR/Cas9システムを用いたノックインフィッシュの作製は、簡便、かつ高効率であり、今後、トランスジェニックフィッシュ作製の標準的な手法の一つとなると予想される。また、他の動物種にも広く応用されることが期待される。
Yukiko Kimura, Yu Hisano, Atsuo Kawahara and Shin-ichi Higashijima
Efficient generation of knock-in transgenic zebrafish carrying reporter/driver genes by CRISPR/Cas9-mediated genome engineering.
Scientific reports 4, 6545 (2014). DOI: 10.1038/srep06545
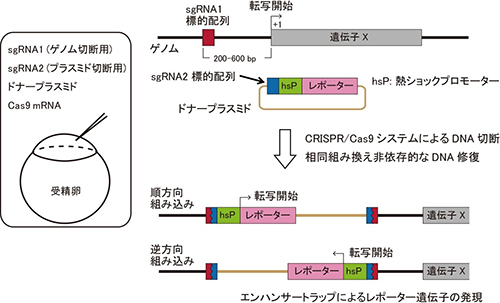 CRISPR/Cas9システムによる標的DNA配列の切断にはヌクレアーゼであるCas9と、標的配列に相同な配列を持ち、Cas9を標的配列に誘導するガイダンス分子となる短鎖RNA(sgRNA)が必要である。ノックインフィッシュ作製の際には、ゲノムを標的とするsgRNA1と、ドナープラスミドを標的とするsgRNA2、sgRNA2の標的配列を持つドナープラスミド、Cas9 mRNAを受精卵に注入した。CRISPR/Cas9システムによってドナープラスミドとゲノムが細胞内で同時に切断されると、直鎖化されたドナープラスミドがゲノム切断部位に、相同組み換え非依存的なDNA修復を介して高効率に組込まれる。本研究では、ドナープラスミドに、熱ショックプロモーターとレポーター遺伝子配列も含ませ、ゲノムのsgRNA標的配列は、標的遺伝子の上流に設計した。このドナープラスミドが標的遺伝子上流のゲノム切断部位に組込まれると、熱ショックプロモーターに、標的遺伝子の制御領域が働きかけ、標的遺伝子発現細胞におけるレポーター遺伝子の発現を促す。
CRISPR/Cas9システムによる標的DNA配列の切断にはヌクレアーゼであるCas9と、標的配列に相同な配列を持ち、Cas9を標的配列に誘導するガイダンス分子となる短鎖RNA(sgRNA)が必要である。ノックインフィッシュ作製の際には、ゲノムを標的とするsgRNA1と、ドナープラスミドを標的とするsgRNA2、sgRNA2の標的配列を持つドナープラスミド、Cas9 mRNAを受精卵に注入した。CRISPR/Cas9システムによってドナープラスミドとゲノムが細胞内で同時に切断されると、直鎖化されたドナープラスミドがゲノム切断部位に、相同組み換え非依存的なDNA修復を介して高効率に組込まれる。本研究では、ドナープラスミドに、熱ショックプロモーターとレポーター遺伝子配列も含ませ、ゲノムのsgRNA標的配列は、標的遺伝子の上流に設計した。このドナープラスミドが標的遺伝子上流のゲノム切断部位に組込まれると、熱ショックプロモーターに、標的遺伝子の制御領域が働きかけ、標的遺伝子発現細胞におけるレポーター遺伝子の発現を促す。